近日,清华大学交叉信息研究院的曾坚阳研究组与北京大学马剑竹课题组、索尔克生物研究所、加利福尼亚大学、上海交通大学合作,首次将基因调控网络显式的引入单细胞RNA测序(scRNA-seq)数据建模中,其效果在调控网络预测、scRNA-seq数据低维嵌入、scRNA-seq数据模拟生成等任务中优于现有方法。
基因调控网络(Gene Regulatory Network, GRN)是研究细胞分化、细胞重编程中的关键问题。近年来,已有工作将统计学方法、自回归机器学习算法应用于这一领域,但这些算法在预测准确性上仍然存在提升空间。随着深度学习在因果推断中的应用,研究人员提出了DeepSEM模型,通过设计GRN layer 和 Inverse GRN layer,首次将深度学习应用于基因调控网络的预测,提高了基因调控网络预测的准确度。
与此同时,基因调控网络作为每一类细胞特有的性质,却被绝大部分scRNA-seq低维嵌入模型所忽视。在本项研究中,研究人员利用GRN layer 和Inverse GRN layer,将被预测的基因调控网络显式引入scRNA-seq 低维嵌入中。在聚类和可视化任务中,DeepSEM在基准数据集上优于现有方法的效果。与此同时,研究人员还进一步将DeepSEM模型应用于scRNA-seq数据模拟生成,在提出的GRN一致性指标上高于现有scRNA-seq 数据模拟生成方法。
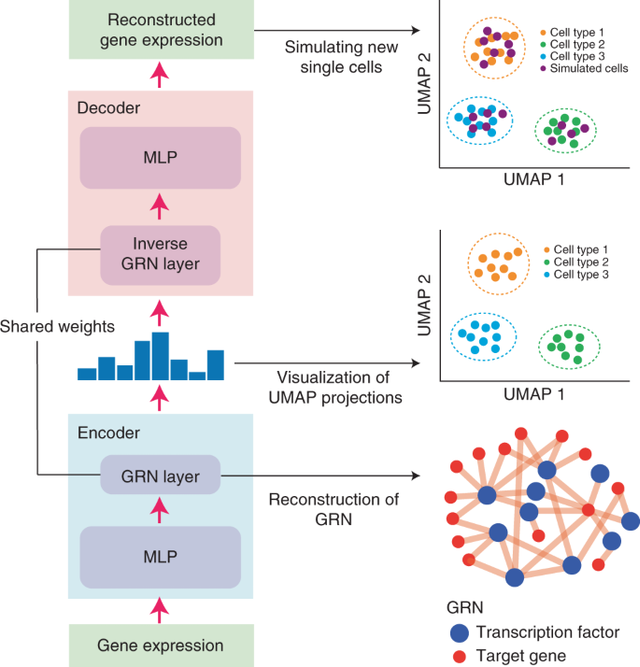
DeepSEM模型框架图
此项研究工作巧妙地将基因调控网络预测和scRNA-seq数据建模进行结合,实现了一个模型、多个用途,为将来scRNA-seq数据分析和计算方法研究提供了新的思路和切入点。
该成果论文“基于神经网络的基因调控网络建模”(Modeling Gene Regulatory Networks Using Neural Network Architectures)发表于《自然·计算科学》(Nature Computational Science)期刊。论文第一作者为清华大学交叉信息院2018级博士生束晗涛,共同通讯作者为北京大学人工智能研究院马剑竹副教授、清华大学交叉信息研究院曾坚阳副教授。该研究得到了国家自然科学基金、南京图灵人工智能研究院的支持。
① 凡本站注明“稿件来源:中国教育在线”的所有文字、图片和音视频稿件,版权均属本网所有,任何媒体、网站或个人未经本网协议授权不得转载、链接、转贴或以其他方式复制发表。已经本站协议授权的媒体、网站,在下载使用时必须注明“稿件来源:中国教育在线”,违者本站将依法追究责任。
② 本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者在两周内速来电或来函联系。





 中国教育在线
中国教育在线



